Funzioni, tipi ed esempi degli enzimi di restrizione
- 4953
- 600
- Silvano Montanari
IL enzimi di restrizione Sono endonucleasi usate da alcuni archi e batteri per inibire o "limitare" la propagazione dei virus all'interno. Sono particolarmente comuni nei batteri e fanno parte del loro sistema di difesa del DNA estero noto come sistema di restrizione/modifica.
Questi enzimi catalizzano il taglio del DNA a doppia banda in luoghi specifici, riproducibili e senza l'uso di energia aggiuntiva. La maggior parte richiede la presenza di cofattori come il magnesio o altri cationi bivalenti, sebbene alcuni richiedano anche ATP o S-adenosil metionine.
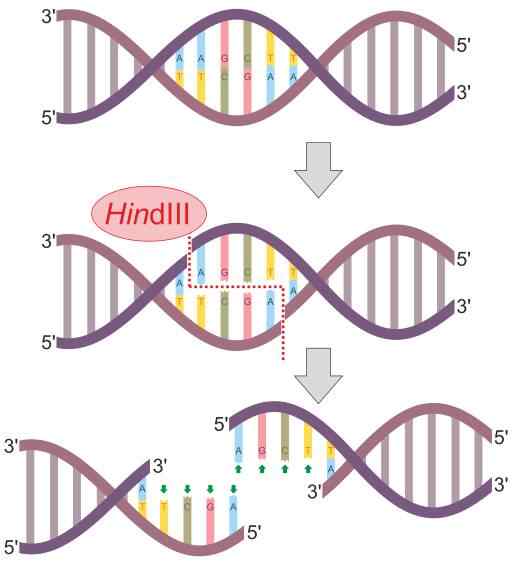
 Schema di reazione enzimatica di restrizione hindii (fonte: Helixitta [CC BY-SA 4.0 (https: // creativeCommons.Org/licenze/by-sa/4.0)] via Wikimedia Commons)
Schema di reazione enzimatica di restrizione hindii (fonte: Helixitta [CC BY-SA 4.0 (https: // creativeCommons.Org/licenze/by-sa/4.0)] via Wikimedia Commons) Le endonucleasi di restrizione furono scoperte nel 1978 da Daniel Nathans, Arber Werner e Hamilton Smith, che ricevette il premio Nobel in medicina per la loro scoperta. Il suo nome generalmente deriva dall'organismo in cui vengono osservati per la prima volta.
Tali enzimi sono ampiamente utilizzati nello sviluppo di metodi di clonazione del DNA e altre biologia molecolare e strategie di ingegneria genetica. Le sue caratteristiche di riconoscimento di sequenze specifiche e capacità di taglio delle sequenze vicine ai siti di riconoscimento li rendono potenti strumenti nella sperimentazione genetica.
I frammenti generati dagli enzimi di restrizione che hanno agito su una particolare molecola di DNA possono essere usati per ricreare una "mappa" della molecola originale utilizzando le informazioni sui siti in cui l'enzima ha tagliato il DNA.
Alcuni enzimi di restrizione possono avere lo stesso sito di riconoscimento nel DNA, ma non lo tagliano necessariamente allo stesso modo. Pertanto, ci sono enzimi che fanno tagli lasciando romos e enzimi che tagliano gli estremi coesivi, che hanno diverse applicazioni in biologia molecolare.
Attualmente ci sono centinaia di diversi enzimi di restrizione disponibili in commercio, offerti da diverse case commerciali; Questi enzimi funzionano come forbici molecolari "su misura" per scopi diversi.
[TOC]
Funzioni
Gli enzimi di restrizione soddisfano la funzione opposta delle polimerasi, poiché sono idrolizzati o rompono il legame estere all'interno del legame fosfodioso tra nucleotidi adiacenti in una catena nucleotidica.
In biologia molecolare e ingegneria genetica sono strumenti ampiamente utilizzati per la costruzione di vettori di espressione e clonazione, nonché per l'identificazione di sequenze specifiche. Sono anche utili per la costruzione di genomi ricombinanti e hanno un grande potenziale biotecnologico.
I recenti progressi nella terapia genica fanno l'attuale uso degli enzimi di restrizione per l'introduzione di geni determinati nei vettori che sono veicoli per il trasporto di tali geni alle cellule viventi e che probabilmente hanno la capacità di inserirsi nel genoma cellulare per eseguire cambiamenti permanenti.
Meccanismo di azione
Gli enzimi di restrizione possono catalizzare il taglio del DNA a doppia banda, sebbene alcuni siano in grado di riconoscere sequenze di DNA a banda semplice e persino RNA. Il taglio si verifica dopo il riconoscimento delle sequenze.
Può servirti: ciclo glioxilato: caratteristiche, reazioni, regolazione, funzioniIl meccanismo d'azione consiste nell'idrolisi del legame fosfodioso tra un gruppo di fosfato e un desossiribosio nello scheletro di ciascun DNA del DNA. Molti degli enzimi sono in grado di tagliare nello stesso posto che riconoscono, mentre altri tagliano tra 5 e 9 coppie di basi prima o dopo lo stesso.
Normalmente questi enzimi tagliati all'estremità 5 'del gruppo fosfato, dando origine a frammenti di DNA con un'estremità fosforile da 5' e un terminale di fine idrossile 3 '.
Poiché le proteine non entrano in contatto diretto con il sito di riconoscimento del DNA, questi devono essere traslocati tempi successivi fino a quando il sito specifico non viene raggiunto, forse attraverso meccanismi "scorrevoli" sul filamento del DNA.
Durante il taglio enzimatico, il collegamento fosfodioso di ciascuno dei fili del DNA è posizionato all'interno di uno dei siti attivi di enzimi di restrizione. Quando l'enzima lascia il sito di riconoscimento e taglio, lo fa attraverso associazioni transitorie non specifiche.
Ragazzi
Attualmente sono noti cinque tipi di enzimi di restrizione. Successivamente, una breve descrizione di ognuno:
Enzimi di restrizione di tipo I
Questi enzimi sono grandi proteine pentamericane con tre subunità, una di restrizione, una metilazione e un'altra per il riconoscimento delle sequenze di DNA. Queste endonucleasi sono proteine multifunzionali in grado di catalizzare le reazioni di restrizione e modifica, hanno ATPASA e anche attività topoisomera del DNA.
Gli enzimi di questo tipo furono le prime endonucleasi da scoprire, furono purificati per la prima volta negli anni '60 e da allora sono stati studiati con grande profondità.
Gli enzimi di tipo I non sono ampiamente utilizzati come strumento biotecnologico, poiché il sito di taglio può essere a una distanza variabile fino a 1.000 coppie di base per quanto riguarda il sito di riconoscimento, che le rende inaffidabili in termini di riproducibilità sperimentale.
Enzimi di restrizione di tipo II
Sono enzimi composti da omodimeri o tetrameri che tagliano il DNA in siti definiti tra 4 e 8 bp di lunghezza. Questi siti di taglio sono in genere palindromici, cioè riconoscono le sequenze che vengono lette allo stesso modo in entrambe le direzioni.
Molti degli enzimi di restrizione di tipo II nel batterio tagliano il DNA quando riconoscono il loro carattere estraneo, poiché non ha le modifiche tipiche che il proprio DNA dovrebbe avere.
Questi sono enzimi di restrizione più semplici poiché non richiedono alcun cofattore diverso dal magnesio (Mg+) per riconoscere e tagliare le sequenze di DNA.
La precisione degli enzimi di restrizione di tipo II nel riconoscere e tagliare sequenze semplici nel DNA in posizioni precise li rende uno dei più utilizzati e indispensabili nella maggior parte dei rami della biologia molecolare.
Può servirti: mutualismo: caratteristiche, tipi, esempiAll'interno del gruppo di enzimi di restrizione di tipo II sono classificati in più sottoclassi in base a alcune proprietà uniche per ciascuna. La classificazione di questi enzimi viene effettuata aggiungendo lettere dell'alfabeto, da a a z che segue il nome dell'enzima.
Alcune delle sottoclassi più conosciute per la loro utilità sono:
Sottoclasse IIA
Sono diversi subunità dímeros. Riconoscono sequenze asimmetriche e sono usati come precursori ideali per la generazione di enzimi di taglio.
Sottoclasse IIB
Sono composti da un altro dimeri e tagliano il DNA su entrambi i lati della sequenza di riconoscimento. Hanno tagliato entrambi i fili del DNA in un intervallo di coppie di basi più tardi dal sito di riconoscimento.
Sottoclasse IIC
Gli enzimi di questo tipo sono polipeptidi con divisione e funzioni di modifica dei fili del DNA. Questi enzimi tagliano entrambi i fili asimmetricamente.
Sottoclasse iie
Gli enzimi di questa sottoclasse sono i più utilizzati nell'ingegneria genetica. Hanno un sito catalitico e generalmente richiedono un effettore di alestro. Questi enzimi devono interagire con due copie della loro sequenza di riconoscimento per effettuare un taglio efficiente. All'interno di questa sottoclasse ci sono enzimi ecorii ed ecori.
Enzimi di restrizione di tipo III
Le endonucleasi di restrizione di tipo III sono composte solo da due subunità, una è responsabile del riconoscimento e della modifica del DNA, mentre l'altro è responsabile del taglio della sequenza.
Questi enzimi richiedono due cofattori per il loro funzionamento: ATP e magnesio. Gli enzimi di restrizione di questo tipo hanno due siti di riconoscimento asimmetrici, traslocano il DNA in modo dipendente dall'ATP e lo tagliano tra 20 e 30 bp adiacenti al sito di riconoscimento.
Enzimi di restrizione di tipo IV
Gli enzimi di tipo IV sono facili da identificare poiché hanno tagliato il DNA con segni di metilazione, sono costituiti da diverse subunità che sono responsabili del riconoscimento e del taglio della sequenza del DNA. Questi enzimi usano cofattori GTP e magnesio bivalente.
I siti di taglio specifici comprendono catene nucleotidiche con residui di citosina metilati o idrossimesity in uno o entrambi i fili di acidi nucleici.
Enzimi di restrizione di tipo V
Questo classificazione raggruppa gli enzimi di tipo CAS croccante, che identificano e tagliano specifiche sequenze di DNA di organismi invasori. Gli enzimi CAS usano una guida sintetizzata del filamento di RNA per il riconoscimento e l'attacco degli organismi invasori.
Gli enzimi classificati come tipo V sono polipeptidi strutturati dagli enzimi di tipo I, II e II. Possono tagliare sezioni di DNA di quasi tutti gli organismi e con una vasta gamma di lunghezza. La sua flessibilità e facilità di occupazione rendono questi enzimi uno degli strumenti più utilizzati nell'ingegneria genetica attualmente insieme agli enzimi di tipo II.
Può servirti: propionibacteriumEsempi
Gli enzimi di restrizione sono stati utilizzati per la rilevazione dei polimorfismi di DNA, in particolare negli studi sulla genetica della popolazione e negli studi evolutivi mediante DNA mitocondriale, al fine di ottenere informazioni sui tassi di sostituzione dei nucleotidi.
Al momento, i vettori utilizzati per la trasformazione di batteri con vari scopi hanno siti multiclonage in cui si trovano siti di riconoscimento per enzimi multipli di restrizione.
Tra questi enzimi i più popolari sono Ecori, II, III, IV e V, ottenuti e descritti per la prima volta da E. coli; Hindiii, da H. Influenzae e Bamhi di B. Amyloliquefaciens.
Riferimenti
- Bikle, t. A., & Kruger, D. H. (1993). Biologia della restrizione del DNA. Recensioni microbiologiche, 57(2), 434-450.
- Boyaval, p., Moineau, s., Romero, d. A., & Horvath, P. (2007). CRISPR fornisce l'acquisizione di virus nei procarioti. Scienza, 315(Marzo), 1709-1713.
- MOTOREell, d. (2002). La prospettiva molecolare: endonucleasi di restrizione. Fondamenti di cellule staminali della medicina del cancro, venti, 190-191.
- Halford, s. E. (2001). Saltare, saltare e looping da enzimi di restrizione. Transazioni della società biochimica, 29, 363-373.
- Jeltsch, a. (2003). Mantenimento dell'identità delle specie e controllo della speciazione dei batteri: una nuova funzione per i sistemi di restrizione/modifica? Gene, 317, 13-16.
- Krebs, j., Goldstein, e., & Kilpatrick, s. (2018). Geni XII di Lewin (12 ed.). Burlington, Massachusetts: Jones & Bartlett Learning.
- Li, e., Pane, s., Zhang, e., Ren, m., Feng, m., Peng, n.,... lei, Q. (2015). Sfisione di sistemi CRISPR-Cas di tipo I e tipo III per l'editing del genoma. Ricerca degli acidi nucleici, 1-12.
- Pretenza, w. A. M., Dryden, d. T. F., Raleight, e. A., & Wilson, G. G. (2013). Enzimi di restrizione di tipo I e loro parenti. Ricerca degli acidi nucleici, 1-25.
- Nathans, d., & Smith, H. O. (1975). Endonucleasi di restrizione nell'analisi e nella ristrutturazione delle molecole di DNA. Annu. Rev. Biochimico., 273-293.
- Nei, m., & Tajima, F. (1981). Polimorfismo del DNA rilevabile da endonucleasi di restrizione. Genetica, 145-163.
- Pingoud, a., Fuxreiter, m., Pingoud, v., & Wende, W. (2005). Scienze della vita cellulare e molecolare di tipo II restrizione endonucleasi: struttura e meccanismo. Scienze della vita cellulare e molecolare CML, 62, 685-707.
- Roberts, r. (2005). Come gli enzimi di restrizione sono diventati i cavalli di lavoro della biologia molecolare. PNA, 102(17), 5905-5908.
- Roberts, r. J., & Murray, K. (1976). Restrizione endonucleasi. Recensioni critiche in biochimica, (Novembre), 123-164.
- Stoddard, b. L. (2005). Struttura e funzione endonucleasi di homing. Recensioni trimestrali di biofisica, 1-47.
- TOCK, m. R., & Dryden, D. T. F. (2005). La biologia della restrizione e dell'anti-restrizione. Opinione attuale in microbiologia, 8, 466-472. https: // doi.org/10.1016/j.Il mio b.2005.06.003
- Wilson, g. G., & Murray, N. E. (1991). Sistemi di limitazione e modifica. Annu. Rev. Genet., 25, 585-627.
- Wu, z., & MOU, K. (2016). Intuizioni genomiche sulla virulenza e la genetica della popolazione di Campylobacter Jejuni. Infec. Dis. Trasgr. Med., 2(3), 109-19.
- Yuan, r. (1981). Struttura e meccanismo di endonucleasi di restrizione multifunzionale. Annu. Rev. Biochimico., cinquanta, 285-315.
- « Generazione di caratteristiche, autori e opere del 1914
- Proprietà ed esempi di eventi reciprocamente esclusivi »

