Meccanismi di replicazione del DNA, in procarioti ed eucarioti
- 4231
- 334
- Silvano Montanari
IL Replica di DNA (Acido desoxyribonucleico) consiste nella copia del genoma, cioè tutte le informazioni genetiche contenute nel DNA di un organismo, per produrre due copie identiche. Il genoma ha le informazioni necessarie per costruire un organismo completo.
Prima della divisione cellulare, si verifica la replicazione del DNA. Attraverso la meiosi, si verificano gameti per la riproduzione sessuale. Attraverso la mitosi, la sostituzione cellulare si verifica (ad esempio, la pelle e il sangue) e lo sviluppo (ad esempio, tessuti e organi).
 Fonte: i, madprime [cc0]
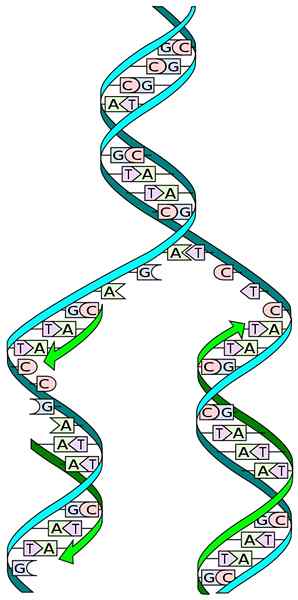
Fonte: i, madprime [cc0] La conoscenza della struttura del DNA ci consente di comprendere il modo in cui si verifica la sua replicazione. La struttura del DNA è costituita da una doppia elica, composta da due catene antipaalli di successivi nucleotidi, le cui basi di azoto sono integrate specificamente.
Durante la replica, ciascuna delle catene della doppia catena del DNA funge da stampo per la biosintesi di una nuova catena. Le due catene appena sintetizzate hanno basi complementari alle basi della catena dello stampo: adenina (a) con timina (t) e citosina (c) con guanina (G).
Diversi enzimi e proteine partecipano alla replicazione del DNA. Ad esempio, aprendo l'elica del doppio DNA, mantenendo il DNA aperto e aggiungendo deossiribonucleosidos-5'-trifosfato (DNTP) per formare la nuova catena.
[TOC]
La replicazione del DNA è semi -conservato
Sulla base della struttura del DNA, Watson e Crick hanno proposto che la replicazione del DNA si verifichi in modo semi -conservato. Ciò è stato dimostrato da Meselson e Stahl segnando il DNA di Escherichia coli Con il pesante isotopo di azoto, quindiciN, seguendo per diverse generazioni il modello di distribuzione in un terreno di coltura con azoto chiaro, 14N.
Messelson e Stahl hanno scoperto che, nella prima generazione, le molecole di DNA delle due figlie, ogni molecola contrassegnata con una catena con il pesante isotopo di azoto e un'altra con l'isotopo della luce. A differenza della molecola di DNA parentale, che aveva le due catene contrassegnate con il pesante isotopo, quindiciN.
Nella seconda generazione, il 50% delle molecole di DNA era come quella della prima generazione e l'altro 50% aveva solo azoto leggero. L'interpretazione di questo risultato è che l'elica a doppia figlia ha una catena parentale (che funziona come stampo) e una nuova catena.
Il meccanismo di replicazione semi -conservato implica la separazione delle catene del DNA e l'accoppiamento di basi complementari per mezzo di nucleotidi successivi, producendo figlie a due figlie doppie.
Replica nelle batterie
Inizio della replicazione del DNA nei batteri
I batteri DNA sono costituiti da un cromosoma circolare e ha solo un sito di origine della replicazione. Da questo sito, la biosintesi delle due figlie avviene in modo bidirezionale, formando due forcelle di replica che si muovono in direzioni opposte all'origine. Alla fine, le forche sono completate la replica.
La replica inizia con l'unione delle proteine DNAA nel sito di origine. Queste proteine a loro volta formano un complesso. Quindi le proteine HU e IHF sono legate.
Successivamente, le proteine DNAC sono unite, che rendono le elicasi del DNA. Questi aiutano a rilassarsi il DNA e rompere i legami idrogeno, formati in coppie di basi. Quindi, le due catene si separano ancora di più, formando due semplici catene.
Può servirti: cos'è la permutazione cromosomica?Topoisomerasi II, o DNA girasa, si muove davanti a DNA Helicy. Le proteine di legame del DNA a catena singola (SSB) mantengono separate le catene di DNA. Pertanto, può iniziare la biosintesi della catena della figlia.
Biosintesi delle figlie catene del DNA nei batteri
L'enzima prima è responsabile della sintesi di catene di RNA corte chiamate primer, che hanno da 10 a 15 nucleotidi di lunghezza. Il DNA polimerasi inizia ad aggiungere 5'-trifosfato (DNTPS) al 3'-OH dello zucchero di innesco, dopo di che la catena continua a crescere alla stessa estremità.
Poiché le catene di DNA sono antiparalleli, un primer viene sintetizzato nella catena guida e molti primer nella catena ritardata. Per questo motivo, la biosintesi della catena ritardata è discontinua. Sebbene le catene del DNA siano antiparalleli, la forcella di replica si muove in una direzione.
La DNA polimerasi è responsabile della formazione di legami covalenti tra nucleotidi adiacenti delle catene appena sintetizzate, in direzione 5'®3 '. In E. coli, Esistono cinque DNA di polimerasi: DNA polimerasi I e III eseguono la replicazione del DNA; e le DNA polimerasi II, IV e V sono responsabili della replica e della replica del DNA danneggiato.
La maggior parte della replicazione è effettuata dalla DNA polimerasi III, che è un oloenzima che ha 10 diverse subunità con diverse funzioni nella replicazione del DNA. Ad esempio, Alpha Subunità è responsabile della creazione di legami tra nucleotidi.
Un complesso enzimatico è responsabile della replicazione del DNA nei batteri
L'elicasi di DNA e prima è unita per formare un complesso chiamato primosoma. Questo si muove lungo il DNA, agendo in modo coordinato per separare le due catene parentali, sintetizzando i primer ogni determinato intervallo sulla catena ritardata.
Il primosoma si lega fisicamente alla DNA polimerasi III e forma il replisoma. Due DNA polimerasi III sono responsabili della replica del DNA della guida e delle catene ritardate. Rispetto alla DNA polimerasi III, la catena ritardata forma un loop, che consente l'aggiunta di nucleotidi a questa catena nella stessa direzione della catena guida.
L'aggiunta di nucleotidi alla catena guida è continua. Mentre nel ritardo è discontinuo. Si formano frammenti di 150 nucleotidi di lunghezza, chiamati frammenti di okazaki.
L'attività di esonucleasi 5 ' -> 3' della DNA polimerasi I è responsabile dell'eliminazione dei primer e del riempimento, aggiungendo nucleotidi. Un ligasi enzimatico sigilla gli spazi tra i frammenti. La replica termina quando le due hoquillas di replica sono in una sequenza di completamento.
La proteina si lega alla sequenza di terminazione, fermando il movimento della forcella di replica. Topoisomerasi II consente la separazione dei due cromosomi.
Il trifosfato depoxiribonucleotidi è usato dalla DNA polimerasi
Il trifosfato di dexinucleoside (DNTP) contiene tre gruppi di fosfato uniti al carbonio 5 'di deossiribosa. I DNTPS (DATP, DTTP, DGTP e DCTP) si uniscono alla catena dello stampo seguendo la regola AT/GC.
Può servirti: plesiomorphyLa DNA polimerasi catalizza la seguente reazione: il gruppo idrossilico (-OH) 3 'del nucleotide della catena in crescita reagisce con l'alfa fosfato del DNTP in arrivo, rilasciando pirofosfato inorganico (PPI) (PPI) (PPI). L'idrolisi PPI produce energia per la formazione del legame covalente, o legame fosfodié, tra i nucleotidi della catena di crescita.
Meccanismi che garantiscono la fedeltà di replica del DNA
Durante la replicazione del DNA, il DNA polimerasi III commette un errore per 100 milioni di nucleotidi. Sebbene la probabilità di errore sia molto bassa, ci sono meccanismi che garantiscono la fedeltà nella replicazione del DNA. Questi meccanismi sono:
1) Stabilità in APAR BASE. L'energia del legame idrogeno tra AT/GC è maggiore rispetto alle coppie di basi errate.
2) Struttura del sito attivo della DNA polimerasi. La DNA polimerasi catalizza preferibilmente i nucleotidi con basi corrette nella catena opposta. Un accoppiamento cattivo delle basi provoca una distorsione dell'elica del doppio DNA, che impedisce al nucleotide errato di occupare il sito attivo dell'enzima.
3) Test di lettura. La DNA polimerasi identifica nucleotidi errati incorporati ed li elimina dalla catena della figlia. L'attività di esonucleasi del DNA della polimerasi rompe i legami di fosfodié tra nucleotidi all'estremità 3 'della nuova catena.
Replicazione del DNA negli eucarioti
A differenza della replica in Prokaryotes, la cui replica inizia in un singolo sito, la replica eucariotica inizia in più siti di origine e la forcella di replica si muove in modo bidirezionale. Successivamente, tutte le forcelle di replica vengono unite, formando due cromatidi sorelle uniti al centromero.
Gli eucarioti hanno molti tipi di DNA polimerasi, i cui nomi usano lettere greche. DNA polimerasi α forma un complesso con prima. Questo complesso sintetizza i primer brevi costituiti da 10 nucleotidi di RNA seguiti da 20 a 30 nucleotidi di DNA.
Quindi DNA polimerasi ε O Δ catalizza l'allungamento della catena della figlia dal primer. DNA polimerasi ε È coinvolto nella sintesi della catena principale, mentre la DNA polimerasi Δ Sintetizzare la catena ritardata.
DNA polimerasi Δ Allungare il frammento di Okazaki sinistro fino a raggiungere il primer RNA destro, producendo un breve sollevamento del primer ("lembo corto"). A differenza dei prokaryotes, in cui un DNA polimerasi elimina il primer, negli eucarioti un enzima di endonucleasi elimina il primer RNA.
Successivamente, una ligasi del DNA sigilla i frammenti di DNA adiacenti. Il completamento della replica avviene con la dissociazione delle proteine della forcella di replica.
IL Replicazione del DNA in eucarioti e ciclo cellulare
La replica negli eucarioti si verifica nella fase s del ciclo cellulare. Le molecole di DNA replicate sono separate in due cellule figlie durante la mitosi. Le fasi G1 e G2 separano la fase S e la mitosi. La progressione attraverso ogni fase del ciclo cellulare è altamente regolata da chinasi, fosfatasi e proteasi.
Nella fase G1 del ciclo cellulare, il complesso di riconoscimento delle origini (OCR) si unisce al sito di origine. Ciò induce l'unione di MCM Helicas e altre proteine, come CDC6 e CDT1, per formare un complesso di pre-replicazione (PRERC). Las Helicase MCM si unisce alla catena guida.
Può servirti: geni collegatiNella fase s, PRERC diventa un sito di replica attivo. Vengono rilasciate le proteine OCR, CDC6 e CDT1 e l'elicasi MCM si muove nella direzione da 3 'a 5'. Una volta terminata la replica, questo verrà riavviato nel ciclo cellulare successivo.
Replica delle estremità dei cromosomi negli eucarioti
Le estremità dei cromosomi sono conosciute come telomeri, che consistono in sequenze ripetute in tandem e di una regione 3 'che eccelle, da 12 a 16 nucleotidi di lunghezza.
La DNA polimerasi non è in grado di replicare l'estremità 3 'delle catene del DNA. Questo perché il DNA polimerasi può sintetizzare solo il DNA nell'indirizzo 5'-3 'e può solo allungare le catene preesistenti, senza essere in grado di sintetizzare un primer in questa regione. Di conseguenza, i telomeri vengono accorciati in ogni round di replica.
L'enzima telomerasi impedisce l'accorciamento dei telomeri. La telomerasi è un enzima che ha subunità proteina e RNA (terzo). Quest'ultimo si lega alle sequenze ripetute di DNA e consente alla telomerasi di unirsi all'estremità 3 'del telomero.
Una sequenza di RNA dietro l'Unione situata come stampo per la sintesi di una sequenza di sei nucleotidi (polimerizzazione) alla fine della catena del DNA. L'allungamento del telomero è catalizzato da subunità di telomerasi, chiamata telomerasi reverse telomerasi (TERT).
Dopo la polimerizzazione, avviene la traslocalizzazione, costituita dal movimento della telomerasi in una nuova estremità della catena del DNA, unendosi ad altri sei nucleotidi fino alla fine.
Le funzioni di altre polimerasi di DNA negli eucarioti
DNA polimerasi β Ha un ruolo importante nell'eliminazione di basi errate del DNA, ma non è coinvolto nella replicazione del DNA.
Molti DNA di polimerasi scoperto appartengono al gruppo polimerasi "replicante translesion. Queste polimerasi sono responsabili della sintesi di catene complementari in una regione di DNA danneggiata.
Esistono diversi tipi di polimerasi "replicante della transleion". Ad esempio, DNA polimerasi η Puoi replicare su Timina Dímeros, che sono prodotti da UV Light.
Replicazione del DNA in archeobatteri
La replicazione del DNA archeobatteri è simile a quella fornita negli eucarioti. Ciò è dovuto alle seguenti: 1) le proteine che partecipano alla replicazione sono più simili a quelle degli eucarioti che a quelli dei procarioti; e 2) sebbene esista un solo sito di replica come nei procarioti, la sua sequenza è simile al sito di origine eucariotica.
La somiglianza nella replicazione tra gli archi e gli eucarioti supporta l'idea che entrambi i gruppi siano filogeneticamente più correlati tra loro rispetto a uno qualsiasi di loro con i prokaryote.
Riferimenti
- Brooker, r. J. 2018. Analisi e principi della genetica. McGraw-Hill, New York.
- Hartwell, l. H., Goldberg, m. L., Fischer, J. A., Cappuccio, l. 2018. Genetica - dai geni dei genomi. McGraw-Hill, New York.
- Kušić-tišma, j. 2011. Aspetti fondamentali della replicazione del DNA. Accesso aperto Intech, Croazia.
- Lewis, r., 2015. Concetti e applicazioni di genetica umana. McGraw-Hill, New York.
- Pierce, b. A. 2005. Genetica - un approccio concettuale. W. H. Freeman, New York.

