Metodi di studio HapLooType, diagnostica, malattie
- 3162
- 208
- Zelida Gatti
UN HaplooType È una regione del genoma che tende a ereditare insieme attraverso più generazioni; In genere, tutto si trova nello stesso cromosoma. Gli aplootipi sono il prodotto del legame genetico e rimangono intatti durante la ricombinazione genetica.
La parola "haplotipo" deriva da una combinazione della parola "aploide" e la parola "genotipo". "Aploide" si riferisce alle cellule con un singolo set di cromosomi e "genotipo" parla della composizione genetica di un organismo.
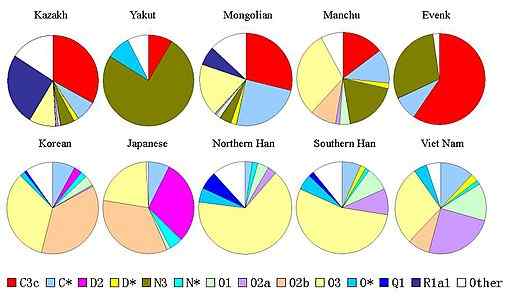
 Schema della distribuzione degli aplotipi cromosomici e nelle popolazioni asiatiche (fonte: Moogalord [dominio pubblico] via Wikimedia Commons) Seguendo la definizione, un aplotipo può descrivere un paio di geni o più che sono ereditati insieme su un cromosoma da uno parentale o può descrivere un cromosoma completamente ereditato da un parentale, come nel caso del cromosoma e degli uomini.
Schema della distribuzione degli aplotipi cromosomici e nelle popolazioni asiatiche (fonte: Moogalord [dominio pubblico] via Wikimedia Commons) Seguendo la definizione, un aplotipo può descrivere un paio di geni o più che sono ereditati insieme su un cromosoma da uno parentale o può descrivere un cromosoma completamente ereditato da un parentale, come nel caso del cromosoma e degli uomini.
Ad esempio, quando gli aplotipi condividono i geni per due diversi personaggi fenotipici, come il colore dei capelli e il colore degli occhi, le persone che hanno un gene del colore dei capelli possiederanno anche l'altro gene per il colore degli occhi.
Gli aplootipi sono uno degli strumenti più utilizzati oggi per lo studio della genealogia, per tracciare l'origine delle malattie, per caratterizzare la variabilità genetica e la filogeografia delle popolazioni di diversi tipi di esseri viventi.
Esistono più strumenti per lo studio degli alooti, uno dei più utilizzati oggi è "Mappa aplotipo“(Hapmap), che è una pagina web che consente di determinare quali sono i segmenti del genoma che sono aplotipi.
[TOC]
Metodi di studio
Gli aplootipi rappresentano un'opportunità per comprendere l'eredità dei geni e il loro polimorfismo. Con la scoperta della tecnica "Reazione a catena della polimerasi" (PCR, inglese "Reazione a catena della polimerasi") Ha avanzato ampiamente nello studio degli aplotipi.
Esistono attualmente numerose metodologie per lo studio degli aplotipi, alcuni dei più punti salienti sono:
Sequenziamento del DNA e rilevamento del polimorfismo a singolo nucleotide (SNP)
Lo sviluppo di tecnologie di sequenziamento di nuove generazioni rappresentava un grande salto per lo studio degli aplotipi. Le nuove tecnologie consentono variazioni fino a una singola base nucleotidica in regioni specifiche di un aplotipo.
Può servirti: DNA ricombinante: tecnica, applicazioni e fondazioniIn bioinformatica, il termine aplotipo viene anche usato per fare riferimento all'eredità di un singolo polimorfismi nucleotidici (SNP) nelle sequenze di DNA.
La combinazione di programmi di bioinformatica con il rilevamento di aplotipi utilizzando il sequenziamento di nuova generazione può essere esattamente identificata la posizione, la sostituzione e l'effetto del cambiamento di ciascuna base sul genoma di una popolazione.
Microsatellites (SSRS)
I microsatelliti o gli SSR derivano dal loro nome dagli inglesi “sImplementa ripetizione E Ripeti in tandem breve". Queste sono sequenze di nucleotidi corti che si ripetono successivamente all'interno di una regione del genoma.
È comune trovare microsatelliti all'interno degli aplotipi non codificanti, quindi, attraverso il rilevamento di variazioni nel numero di ripetizioni di microsatelliti, si possono osservare diversi alleli negli aplotipi degli individui.
Sono stati sviluppati marcatori molecolari di microsatellite per il rilevamento di infiniti aplotipi, dal sexato di piante come la papaia (Carica Papaya) fino al rilevamento di malattie umane come l'anemia falciforme.
Polimorfismi dei frammenti amplificati (AFLP)
Questa tecnica combina l'amplificazione con le reazioni PCR con la digestione del DNA con due diversi enzimi di restrizione. La tecnica rileva loci polimorfici negli aplootipi secondo i diversi siti di taglio nella sequenza del DNA.
Per illustrare meglio la tecnica, immaginiamo tre frammenti di tessuto della stessa lunghezza, ma tagliati in luoghi diversi (questi frammenti rappresentano tre frammenti di aplotipi amplificati usando la tecnica PCR).
All'epoca il tessuto viene tagliato, verranno ottenuti molti pezzi di diverse dimensioni, poiché ogni panno viene tagliato in luoghi diversi. Quando ordiniamo i frammenti in base al tipo di tessuto da cui provengono, possiamo osservare dove si trovano le differenze tra tessuti o aplotipi.
Diagnostica e malattie
Un importante vantaggio dello studio genetico degli aplotipi è che questi rimangono quasi intatti o inalterati per migliaia di generazioni, e ciò consente l'identificazione di antenati remoti e ciascuna delle mutazioni con cui gli individui contribuiscono allo sviluppo delle malattie.
Può servirti: variazioni fenotipicheGli aplootipi nell'umanità variano a seconda delle razze e, a partire da questo scoop, i geni sono stati rilevati all'interno di aplotipi che causano gravi malattie in ciascuna delle razze umane.
Nel progetto Hapmap Sono inclusi quattro gruppi razziali: europeo, yoruba nigeriana, cinese Han e giapponese.
In questo modo, il progetto Hapmap Può coprire i diversi gruppi di popolazioni e tenere traccia dell'origine e dell'evoluzione di molte delle malattie ereditarie che colpiscono ciascuna delle quattro razze.
Una delle malattie che viene diagnosticata più frequentemente usando l'analisi dell'aplotipo è l'anemia falciforme nell'uomo. Questa malattia viene diagnosticata monitorando la frequenza degli aplotipi africani in una popolazione.
Essere una malattia originaria dell'Africa, l'identificazione di aplotipi africani nelle popolazioni ti consente di tracciare facilmente le persone che possiedono la mutazione nella sequenza genetica per le beta globinas degli eritrociti sotto forma di HOZ (caratteristica della patologia).
Esempi
Con gli aplotipi alberi filogenetici che rappresentano le relazioni evolutive tra ciascuno degli aplotipi trovati in un campione di molecole di DNA omologa o nella stessa specie, in una regione che ha una ricombinazione poca o nessuna.
Uno dei rami più studiati attraverso gli aplotipi è l'evoluzione del sistema immunitario umano. Gli aplootipi che codificano per il recettore a pedaggio (un componente chiave del sistema immunitario innato) sono stati identificati per il genoma di Neanderthal e Denisovanos.
Ciò consente loro di tracciare il modo in cui le sequenze genetiche sono cambiate nelle popolazioni di umani "moderni" rispetto alle sequenze di aplotipo che corrispondono agli umani "ancestrali".
Costruire una rete di relazioni genetiche dall'aplotipo mitocondriale.
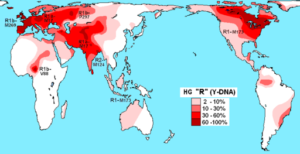 Distribuzione aplogruppo R (DNA-Y) nelle popolazioni native (fonte: Maucioni [CC di 3.0 (https: // creativeCommons.Org/licenze/di/3.0)], via Wikimedia Commons) La diversità degli aplotipi viene utilizzata per tracciare e studiare la diversità genetica degli animali allevati in cattività. Queste tecniche sono utilizzate appositamente per le specie difficili per monitorare la fauna selvatica.
Distribuzione aplogruppo R (DNA-Y) nelle popolazioni native (fonte: Maucioni [CC di 3.0 (https: // creativeCommons.Org/licenze/di/3.0)], via Wikimedia Commons) La diversità degli aplotipi viene utilizzata per tracciare e studiare la diversità genetica degli animali allevati in cattività. Queste tecniche sono utilizzate appositamente per le specie difficili per monitorare la fauna selvatica.
Le specie animali come squali, uccelli e grandi mammiferi come giaguari, elefanti, tra gli altri, sono costantemente valutate geneticamente attraverso aplotipi mitocondriali per monitorare lo stato genetico delle popolazioni in cattività.
Riferimenti
- Bahlo, m., Stankovich, j., Velocità, t. P., Rubio, j. P., Burfoot, r. K., E piede, s. J. (2006). Detezione di dati di aplotipo ampio del genoma mediante SNP o dati di aplotipo di microsatellite. Genetica umana, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). L'introgresso degli aplotipi di Neanderthal-anso-ansovan contribuiscono alla variazione adattiva nei ricevitori a pedaggio umano. The American Journal of Human Genetics, 98 (1), 22-33.
- Da vries, h. G., Van der meulen, m. A., Rozen, r., Halley, d. J., Scheffer, h., Leo, p.,... e tu Meerman, G. J. (millenovecentonovantasei). Identità aplotipo tra individui che condividono un allele di mutazione CFTR. Genetica umana, 98 (3), 304-309
- Deglutire, m. A., Leaver, a. L., Cristiani, f. T., Witt, c. S., Abramo, l. J., & Dawkins, R. L. (1992). Aplotipi ancestrali: popolazione conservatrice MHC aplotipi. Immunologia umana, 34 (4), 242-252.
- Compagni, m. R., Hartman, t., Hermelin, d., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, giugno). Inferenza di aplotipo vincolato dai dati plausibili dell'aplotipo. Nel simposio annuale sulla corrispondenza del modello combinatorio (PP. 339-352). Springer, Berlino, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, j. M., Roy, j., Blumenstiel, b.,... & liu-cordero, s. N. (2002). La struttura dei blocchi di aplotipo nel genoma umano. Science, 296 (5576), 2225-2229.
- Consorzio internazionale HAPMAP. (2005). Una mappa aplotipo del genoma umano. Nature, 437 (7063), 1299.
- Wynne, r., & Wilding, C. (2018). Diversità di aplotipi di DNA mithocondriale e origine degli squali tiger di sabbia in cattività (Carcharias taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo, e. J., Tang, j., Kaslow, r. A., & Zhang, K. (2007). Inferenza di aplotipo per i dati del genotipo presente-abente utilizzando aplotipi e modelli di aplotipo identificati in modo prevalente. Bioinformatica, 23 (18), 2399-2406.
- Young, n. S. (2018). Anemia aplastica. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Caratteristiche dell'eredità olanda, funzioni di geni, degenerazione
- Lentén Caratteristiche, habitat, proprietà, cure »

